- 들어가기 전에
첫 포스팅에서 말했다 시피, 이 포스팅은 전격 반말 포스팅으로 진행됩니다! - 생물학적 지식은 아직도 밝혀지고 있는 만큼, 제가 잘못 쓴 용어와 내용이 많을 수 있습니다. 언제든 지적은 겸허히 받아들이겠습니다!
Transcription : Prokaryote (2. factor-DNA binding)
오랜만이네! 지난 포스팅에서는 RNA polymerase가 어떻게 이루어져 있으며, 그 중 factor를 제외한 core enzyme은 기본적인 RNA synthesis 능력만 가지고 있다는 사실을 말했어. 뿐만 아니라 ‘바로 그 근처에서’ RNA synthesis가 일어나야 한다는 신호를 보내는 녀석이 factor라는 사실을 밝혀낸 실험도 두 가지를 살펴 보았지. 오늘 할 이야기는, 아래의 두 가지 정도야.
- 어디에 붙는가? : -35, -10 box
- 어디랑 붙는가? : factor의 4.2 region, 2.4 region
어떻게 ‘시작’하는지에 대해 정말 자세히 밝혀져 있지? 안정하게 결합하고 있는 DNA를 억지로 떼어낸 다음, 무질서하게 흩어져 있는 여러 조각들을 모아서 질서있는 RNA를 만들어 내야하는, 어떻게 보면 ‘엔트로피가 감소하는’ 비자발적인 과정이 일어나고 있는거지. 그래서 어떻게 transcription을 시작하는지(initiation)에 대한 연구가 가장 많이 이루어져 있다고 받아들여도 돼!
0. 숫자?!
시작하기 전에 짚고 넘어가야 할 부분이 있어. 바로 숫자를 붙이는 방법과 위, 아래 방향성에 대한 거야. DNA에 어떻게 숫자를 붙이느냐하면,
-
Transcription이 시작하는 바로 그곳의 염기(A, G, C, T중 하나!)를 +1이라고 함
-
이 때 주의할 점은, 많이들 ‘start codon’이라고 알고 있는 ATG일필요는 없다는 거! 그건 translation이 시작하는 그 곳의 염기(Met)이고, transcription이 시작하는 위치와는 개념이 달라. transcription은 되는데 translation은 되지 않는 부분들도 당연히 있을 수 있고, 그 부분은 ‘UnTranslated Region(UTR)’라고 불러. 이에 대한 얘기는 나중에 더 해볼게! 아래 그림을 참고해봐!

그림 출처
transcription은 template strand를 기준으로, 5’->3’방향으로 이루어져. 그렇기 때문에 template strand를 기준으로 upstream과 downstream을 결정하게 되는 거야. transcription이 일어나는 방향을 downstream이라고 부르고, 그 반대쪽을 upstream이라고 약속했지. 조심할 것은, Template strand를 기준으로 생각해야 한다는 것! 아래 그림과 같은 경우에는 위쪽 가닥(template strand)의 왼쪽이 3’이니까, 반대로 뒤집어 생각해야 할 거야.
그림 출처
그런데 이상하게도, ‘0’이라는 숫자가 나타내는 염기는 아무것도 없어. +1위치에서 downstream쪽으로 나가면 숫자가 +2, +3,…. 하면서 커질 것은 당연한 건데, upstream쪽으로 나가면 +1 이전의 염기는 ‘-1’으로 표기해. 이건 그렇게 약속을 했기 때문이야.
0-1. 그리고….
아래 내용을 진행하기 전에 꼭 생각해둬야 하는 건, factor는 단백질 상태로 promoter region의 DNA 염기 서열에 결합한다는 사실이야. 그래서, 단백질이 DNA의 염기 서열과 상호작용하고 있다는 사실을 꼭 기억하고 있어야해! 아래에서 계속 얘기할 factor의 region이라는 건, 이미 translation까지 끝난 단백질의 region을 의미하고 있다는 걸 꼭! 혼동을 방지하기 위해 미리 설명 해두고 갈게 ㅎㅎ 그림을 참조!

그림 출처
1. 어디에 붙는가? : -35, -10 box
transcription 이 시작하는 바로 그! 지점보다 조금 더 앞쪽(upstream)에서 열심히 RNA polymerase에게 러브콜을 보내고 있는 녀석들을, Promoter region 이라 부르기로 약속했어. 그 중에서도 factor가 인식하는 두 부분을 발견했지.
왜 갑자기?
사실 왜 갑자기 거기에 붙는다고 생각을 할 수 있었는지 뜬금없을 수도 있어. 시작 지점보다 뒤쪽에서(downstream) 붙어서 조절을 할 수도 있는 노릇이고, 바로 그 지점에 붙을 수도 있잖아. 이 사실을 알기 위해 과학자들은, 그 당시 해독이 끝나있던 단백질들의 유전자 서열들을 쫙~펼쳐놓은 다음, 그 서열들 중에서 공통된 서열을 찾아나가기 시작했어. 그러다 보니, 거의 모든 중요한 단백질을 만드는 유전자들의 개시코돈(AUG) 앞 쪽에 똑같이, 공통적으로 존재하는 서열이 있더라는 거야. 그걸 consensus sequence라고 부를건데, 마침 그런 서열이 아래 그림과 같은 위치에 두 군데에 있더라는거지!
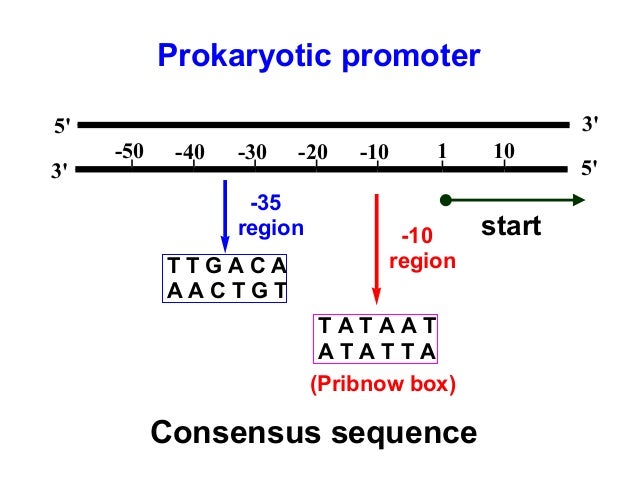
그림 출처
-35 위치에 있는 녀석의 서열은 TTGACA, -10 위치에 있는 녀석의 서열은 TATAAT로 밝혀졌어. 그러면 정말로 여기에 factor가 붙는지 궁금할거 아니야? 그래서 몇 가지 검증을 위한 실험을 더 진행했어. 수많은 실험과 검증이 있었지만, 책에서 소개하고 있는 대표적인 확실한 증거 실험 두 가지만 소개하고 넘어가보려고 해. 이 얘기는 바로 아래에서 계속 진행해볼게!
2. 어디랑 붙는가? : factor의 4.2 region
사실 아래에서 얘기할 두 실험이 진행 되었을 때는 factor 염기서열의 어느 부분이 -35 box, -10 box와 결합하는지에 대한 지식이 조금 있던 상태였어. “아 대충 이렇게 결합할 것 같은데 어떻게 좀 더 확실하게 보여볼까….” 정도의 상태였던 거지. factor를 만드는 유전자 서열을 조각조각 낸 다음 얻어진 단백질들을, -35, -10 box와 결합시켜보는 수많은 노력 끝에 얻어진 결과였던 거야. 그렇게, -35 box와 결합하는 부분은 4.2 region이고, -10 box와 결합하는 부분은 2.4 region이라는 사실을 예측했지.
첫 번째 실험
첫 번째 확실한 증거는, ‘compensationing mutation’이었어. -10 box의 서열을 바꾸면(즉, mutation) 당연히 factor의 2.4 region과의 결합을 안하고, 결과적으로 transcription도 일어나지 않겠지. 이 사실을 역발상을 통해 실험에 활용했어. -10 box의 서열을 바꿔놓고, 바뀐 서열과 상호작용을 잘 하는 다른 단백질이 factor의 2.4 region에 오도록 한 번 더 mutation을 시켜준 거야. 정말 둘이 상호작용을 하고 있으면 이렇게 mutation-mutation을 주었을 때 다시 transcription이 제대로 진행될 거라고 생각한거지. 바로 이 방법, 즉 첫 번째 mutation을 억제하기 위한 또 다른 mutation을 주는 방법을 compensating mutation, 혹은 suppressor mutation이라고 말해.
결론적으로는, -10box와 -35box에 대한 compensating mutation을 각각 factor 2.4, 4.2에 먹였더니 줄었던 transcription level 이 다시 늘어나더라!는거야. 이렇게 factor가 promoter의 어느 위치와 상호작용을 하는지에 대한 증거를 얻었어.
두 번째 실험
두 번째 실험은 좀 더 대놓고 상호작용을 관찰한 실험이야! 하지만 -10 box에 대한 얘기는 없다는 것이 조금 아쉬울 뿐 ㅠ….
filter-binding assay라는 방법으로 한 실험인데, 일단 간단히 원리를 설명하고 넘어갈게! 아마 다음 포스팅에서도 엄청 많이 나올테니까, 제대로 이해해두고 넘어가자구~
- nitrocellulose filter
이 filter는, double-strand로 존재하는 DNA는 통과시키고 단백질이나, 단백질+DNA complex는 통과시키지 못하는 filter야. - labeling with radioisotope
DNA 염기 서열 자체에 radioactive한 동위원소(여기서 한 얘기를 잘 기억한다면 이것이 P라는 걸 알 수 있겠지?)를 처리한 다음, factor의 4.2 region과 충분히 반응을 시켜. 그러면 그 단백질에 radioactivity를 가지는 DNA가 붙어 있을 거야. 그림을 참고하면 좋아!

그림 출처
- filter-binding
우리가 측정할 것은, filter의 radioactivity야. 그런데, 위에서 처리한 factor의 4.2 region-DNA complex를 그대로 nitrocellulose filter에 통과시켜버리면 그냥 filter에 radioactivity가 와글와글하게 있는 결과밖에 안나오겠지? 그래서, 충분한 시간이 흐른 뒤에 다시 radioactivity를 가지지 않는 DNA를 왕창 넣어줘. 그리고 나서 그 시점부터 다시 filter에 통과를 시켜주기 시작하는거지. 그러면서 filter의 radioactivity를 측정하는거야.
종이에 그려가면서 차근차근 이해를 하면……될거야! ㅋㅋㅋㅋ 어쨌든 결론적으로는, factor가 확실히 -35 box와 결합한다면, 시간이 갈 수록 filter의 radioactivity가 감소하는 양상을 보일거야. 반면 결합을 안한다면, 시간이 갈 수록 filter의 radioactivity가 감소하는 정도가 훨씬 덜 하겠지. 그러니까 그래프의 기울기가 급할 수록 결합을 할 확률이 높다는 뜻인거야.
좀 더 정확한 결과를 얻기 위해 몇 가지 실험을 더 했어. factor의 4.2 region을 지운 상태에서 같은 실험을 반복해보기도 했고(아래 그림의 왼쪽 그래프), DNA의 -35 box를 없애거나 -10 box를 없앤 상태에서 같은 실험을 또 해보기도 했지(아래 그림의 오른쪽 그래프).

그림출처 - R. Weaver, Molecular Biology, 4th Ed., Chapter 6
왼쪽 그래프에서, pTac이 의미하는 것이 아무것도 지우지 않은 상태고, P가 factor의 4.2region을 지운 상태야. 4.2 region을 지우니까 확실히, 결합하는 정도가 낮아지네.
또한, 오른쪽 그래프에서 -10 box를 지웠을 때( 10)는 엄청난 속도로 줄어드는데, -35를 지웠을 때( 35)는 결합력이 약해져.
이 두 결과를 통해 유추해보았을 때, factor의 4.2 region은 DNA promoter 상의 -35box와 상호작용을 한다는 결론을 얻는 거야!
결론
- factor는 DNA promoter의 -35 box와 -10 box에 결합한다.
- -35 box에는 factor의 4.2 region이, -10 box에는 factor의 2.4 region이 결합한다.
- compensationing mutation 실험
- nitrocellulose filter binding assay 실험
다음 시간에는
어디에, 어디랑 붙는지에 대해 알았으니 이제 붙은 후에 일어나는 일들을 알아보려고 해. factor가 붙은 후 형성되는 open promoter complex에 대한 이야기도 해보고, factor는 initiation에만 관여를 한 후 일부가 떨어져 나와서 recycling 된다는 사실까지 다뤄보면서 자연스럽게 elongation 으로 넘어가볼…계획이야! 이번에는 좀 더 금방 볼 수 있길 바라며, 다음 포스팅에서 만나!
'정기연재 - 화학생물공학 > [센트럴 도그마] 참새가 들려주는 생명공학' 카테고리의 다른 글
| 2-1. Bacterial transcription (1. sigma factor) (0) | 2016.01.25 |
|---|---|
| 2-1. Bacterial transcription (0. Outline) (0) | 2016.01.06 |
| 1-2. DNA의 구조 (3) (3) | 2015.12.20 |
| 1-2. DNA의 구조 (2) (4) | 2015.11.23 |
| 1-2. DNA의 구조 (1) (2) | 2015.10.21 |





댓글